基于学习转换的一次性医学图像分割中的数据扩增
摘要 图像分割是许多医学应用中的一项重要任务 。基于卷积神经网络的方法获得了最先进的精度;然而 , 它们通常依赖于带有大量的标记数据集的监督训练 。标记医学图像需要大量的专业知识和时间 , 而传统的手工调整的数据扩增方法无法捕捉这些图像中的复杂变化 。
本文提出了一种用来合成有标记的医学图像的自动数据扩增方法 。 本文通过分割磁共振成像(MRI)脑扫描(图像)的任务展示了本文的方法 。本文的方法只需通过一次分段扫描 , 并用一种半监督学习方法对其他无标记扫描(图像)产生影响 。本文从图像中学习一个转换模型 , 并使用该模型与标记样本一起合成扩增标记样本 。每个变换都由一个空间变形场和一次强度变化组成 , 从而能够综合复杂的效应 , 如解剖和图像采集过程的变化 。本文认为用这些新样本来训练一个有监督的分割器 , 比目前最先进的一次性生物医学图像分割方法提供了显著的改进 。
1.导言 语义图像分割对于许多生物医学成像应用是至关重要的 , 如人口分析、诊断疾病和治疗规划 。 当有足够的标记数据可用时 , 基于监督的深度学习分割方法会产生最先进的结果 。 然而 , 手动获得医学图像的分割标签需要相当多的专业知识和时间 。 在大多数临床图像数据集中 , 几乎没有手动标记的图像 。 有限的标记数据的问题由于机器和机构之间图像采集程序的差异而加剧 , 这可能会在分辨率、图像噪声和组织结构的外观方面产生广泛的变化 。
为了克服这些挑战 , 许多有监督的生物医学分割方法侧重于手工设计的预处理步骤和架构 。 使用手工调整的数据扩增来增加训练样本数量也是常见的 。随机图像旋转或随机非线性变形等数据扩增函数易于实现 , 在某些设置中有效地提高了分割精度 。然而 , 这些函数在模拟实际变化方面的能力有限 , 并且对参数[25]的选择非常敏感 。
本文通过学习综合多样化的真实标记样本来解决有限标记数据的挑战 。 本文的新的自动扩增方法是利用未标记的图像 。 使用基于学习的注册方法 , 本文对数据集中图像之间的空间和外观转换集进行建模 。 这些模型捕捉了未标记图像中的解剖和成像多样性 。 本文通过采样转换来合成新的样本 , 并将它们应用于单个标记样本 。
本文证明了本文的方法在脑磁共振成像(MRI)扫描的一次分割任务中的效用 。本文使用本文的方法合成新的标记训练样本 , 使有监督分割网络的训练成为可能 。该策略优于目前最先进的一次性生物医学分割方法 , 包括单 atlas 分割和手工数据扩增下的监督分割 。
图 1:生物医学图像在解剖、对比和纹理(顶部一行)方面经常变化很大) 。与其他一次性分割相比 , 本文的方法能够更准确地分割解剖结构 方法(底部行) 。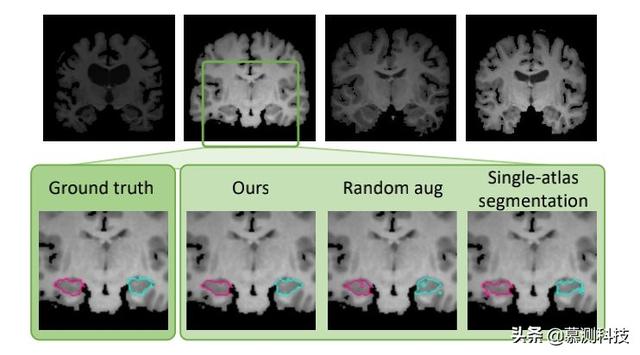 文章插图
文章插图
2.相关工作2.1 医学图像分割
本文专注于脑 MR 图像的分割 , 这是具有挑战性的工作 。 首先 , 人类大脑[28,59,76]表现出大量的解剖性变异 。 其次 , MR 图像强度因特定于缝合线的噪声、扫描仪协议和质量以及其他成像参数的[45]不同而不同 。这意味着组织类可以在不同的图像中 , 甚至是相同 MRI 模式的图像中以不同的强度出现 。
许多现有的分割方法依赖于扫描预处理来减轻这些与强度相关的挑战 。 预处理方法可能运行成本高昂 , 开发现实数据集的技术是一个活跃领域 。 本文的扩增方法从另一个角度解决了这些与强度相关的挑战:它使分割方法对 MRI 扫描中的自然变化具有鲁棒性 , 而不是去除强度变化 。
大量的经典分割方法使用基于 atlas 或 atlas 引导的分割 , 其中标记的参考体积或 atlas 使用变形模型与目标体积对齐 , 标签使用相同的变形[6,13,22,32]传播 。当多个 atlas 可用时 , 它们每个对齐到一个目标卷积 , 扭曲的 atlas 标签被融合[36,41,68,78] 。 在基于 atlas 的方法中 , 受试者之间的解剖变异被一个变形模型捕获 , 并且使用预处理扫描或强度鲁棒性度量(如归一化互相关)来减轻强度变化的挑战 。然而 , 组织外观的模糊性(例如 。不清楚的组织边界、图像噪声)仍然会导致不准确的记录和分割 。 本文通过在不同的现实例子上训练分割模型来解决这一限制 , 使分割器对这种歧义更加健壮 。 本文专注于拥有一个单一的 atlas , 并证明本文的策略优于基于 atlas 的分割 。 如果有多个分段样本可用 , 则可为本文方法所用 。
生物医学分割的监督学习方法近年来得到了广泛的应用 。 为了减少对大量标记训练数据集的需求 , 这些方法通常使用数据扩增以及手工设计的预处理步骤和架构[2,40,53,57,63,65,82] 。
- wmv|怎么把mp4转wmv?转换视频格式,这样操作很掂
- 用于|用于半监督学习的图随机神经网络
- 科技成果|“基于第三代半导体光源的低投射比投影仪关键技术”通过科技成果评价
- 今日|“舜网”学习强国号今日上线 济南报业全媒体矩阵再添新成员
- SK|SK电讯推出自研AI芯片SAPEON X220 深度学习计算速度是常用GPU 1.5倍
- 效果|这个让你相见恨晚的技巧,能让PPT排版更加有设计感,推荐学习
- 如何基于Python实现自动化控制鼠标和键盘操作
- 学习C语言的软件,就突然被我绿了?
- 学习python第二弹
- 喵喵机错题打印机P1:随时打印,随时学习,快速整理错题
