Nature通讯:基于先验知识的网络推断( 二 )
 文章插图
文章插图
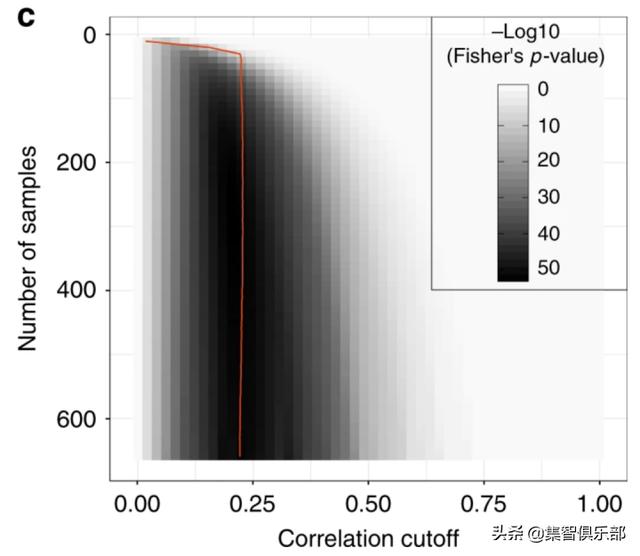
图6:不同大小的样本下的最优截断
为了评估先验知识方法获得的最优网络 , 是否取决于样本大小 , 研究者对部分样本的网络进行优化 。 结果如图所示 , 即使样本量不足200 , 仍然可以获得跟大样本(669个)相同的最优网络 。 这意味着 , 无论样本数据集是大还是小 , 基于先验知识来得到的相关性的最优截断 , 都可以推断出相同的最优网络 。 该方法受样本量规模的影响远远小于传统统计方法 。
3.先验知识质量不同 , 网络推断结果依旧现实生活中很难获得准确且完备的先验知识——因为很难彻底了解许多生物调控系统的分子网络 , 所以仍要用有限的先验知识去解决问题 。 研究者假设 , 只要已知足够多正确的生物学知识 , 即使这些知识粗糙、不完整 , 或者有部分错误 , 该方法也能适用 。 研究者进一步分析了在不同程度的连边缺失、连边出错以及信息模糊情况下的该方法的重构效果 。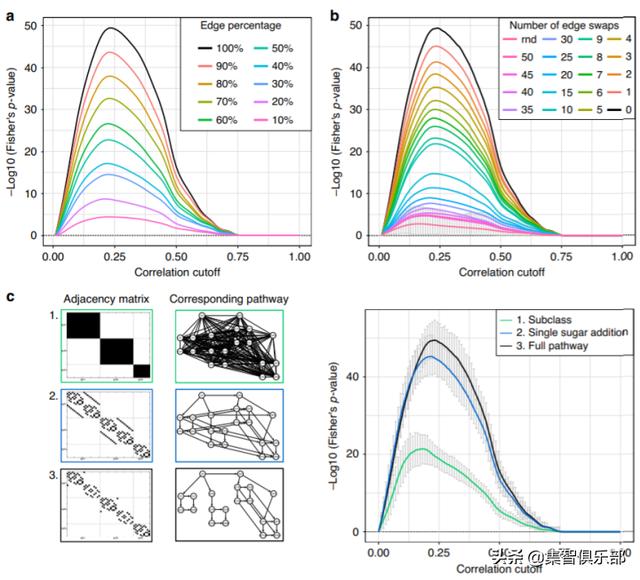 文章插图
文章插图
图7 先验知识质量不佳时的重构结果
研究者从IgG糖基化途径中 , 选择一定比例(从10%到90%不等)的边 , 随机构建网络;还虚构了不同数量的错误连边 , 构建网络 。 但如图7a、7b所示 , 尽管先验知识不足 , 或有部分错误 , 但最优截断点却几乎不受影响 。 研究者还将参考网络进行不同程度的粗粒化 , 如图7c所示 , 发现最优截断点仍然很接近 , 截断的网络是相似的 。 可以发现 , 在先验知识质量较低时 , 该方法依然可以给出较为准确的截断 。 之后 , 研究者还进一步证明了该方法推广到代谢组学和转录组学数据中 , 均有较好的结果 。
4.前景以往的网络推断方法都需要定义一个关键参数阈值p , 由它决定哪些分子间相互作用应该包含在网络中 , 哪些应该舍弃 。 关联网络的推断常常依赖于p的取值 。 这项研究则提出了一种利用先验知识辅助确定关联网络阈值的截断或取值的网络推断/网络重构方法 , 并且证明其在生物领域具有广泛的适用性 。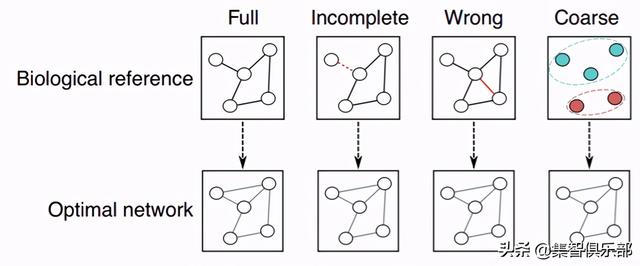 文章插图
文章插图
图8:先验知识质量不佳 , 仍可以推断出最优网络该方法在先验知识不完整、错误或仅提供较模糊的节点分类的情况下都可以较好地推测最优网络结构 , 未来可能成为一个有价值的工具 , 在难以实验证实的情况下推断潜在的生物相互作用 。
作者:刘培源、陈昊
审校:王力飞
【Nature通讯:基于先验知识的网络推断】编辑:邓一雪
- 余额|中兴通讯:现有资金余额仅能确保公司当前经营规模下现金流安全
- 科技成果|“基于第三代半导体光源的低投射比投影仪关键技术”通过科技成果评价
- Nat. Mater:它的出现是否能引领5G通讯的发展?
- 如何基于Python实现自动化控制鼠标和键盘操作
- 需要更换手机了:基于手机构建无人驾驶微型汽车
- 2021年 中兴通讯终端业务将全面回归中国2C市场
- 蓝鲸专访|水滴CTO邱慧:基于业务场景做技术创新,用户需求可分析并唤醒
- 中兴通讯高调官宣:统一操盘中兴、努比亚、红魔三大品牌
- GPU|干货|基于 CPU 的深度学习推理部署优化实践
- 怎么选择合适的无线通讯技术?
