Nature通讯:基于先验知识的网络推断
 文章插图
文章插图
导语
关联网络(correlation network)常常被用于提取生物组学中的相互作用 , 但其连边选择通常基于统计显著性 , 可能无法有效捕捉到生物学机制 。
近日Nature communications上刊载了德国慕尼黑亥姆霍兹中心多位学者的论文“一种利用先验知识选择关系网络截断的策略” 。 文章指出 , 先验知识可以有效地辅助关联网络重构 。 甚至当先验知识粗糙、有缺失、有错误时 , 该方法仍然有效 。
论文题目: A strategy to incorporate prior knowledge into correlation network cutoff selection 论文地址: 文章插图
文章插图
在网络推断(network inference)风潮下 , 利用大数据重构生物网络 , 已经成为生命科学领域的研究热点 。 例如从生物组学大数据中提取出生物分子之间的相互作用 , 进而预测未知的生物反应机制 。
1.传统方法与先验知识辅助方法典型的网络推断工作流程 , 首先是根据预处理的数据 , 估计变量之间的关系 , 形成相关矩阵 。 然后根据假设检验来判断哪些相关关系是显著的 。 最后把显著的相关关系构建成网络(network representation) , 其中节点是数据集中的变量 , 连边代表相关关系 。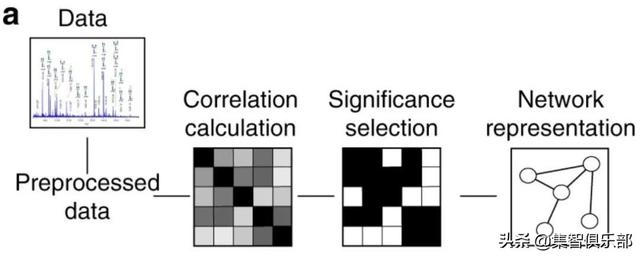 文章插图
文章插图
图1:网络推断工作流
传统方法需要利用假设检验来确定哪些相关系数在统计上是显著的 , 该检验会产生与每个相关系数相关的p值 , 并与给定的显著性水平阈值进行对比 。 只有当相关系数统计显著时 , 才认为其对应连边存在 。 这样的网络推断过程虽然简单 , 却有明显缺陷 , 以至于降低网络推断的鲁棒性和可重复性 。 首先 , 样本量的增大 , 会明显影响统计结果 , 例如样本量越大则网络越稠密 。 其次 , 不同的检验方法有不同的基本假设 , 可能会得到完全不同的网络 。 这些网络尽管在统计学上没问题 , 却未必能有效表示潜在的生物机制 。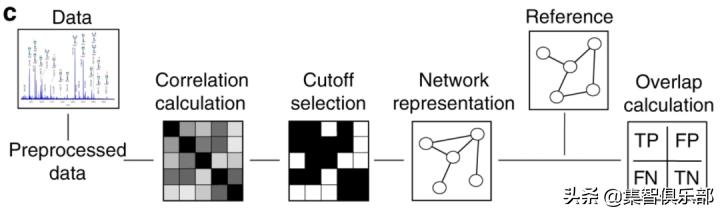 文章插图
文章插图
图2:基于先验知识的网络推断工作流
为了更好地判断相关系数的显著条件 , 即产生连边的阈值 , 从而提升网络推断质量 , 研究者引入了先验知识 。 研究者首先利用先验知识 , 将已知的一定存在关联的节点进行连接 , 构建了参考网络(reference network) 。 然后 , 在对比不同阈值下的(推断的)网络与利用先验知识构建的辅助(参考)网络之间的重叠程度后 , 选择重叠度最好的情况作为网络推断结果 。 重叠度由真阳性(同时出现在关联网络与参考网络)、假阳性(只在关联网络有)、真阴性、假阴性按 Fisher 精确检验方法计算 。 从关联网络中 , 搜索出与参考网络重叠程度最大的网络 , 并截取该网络 , 即为最优网络(optimal network) 。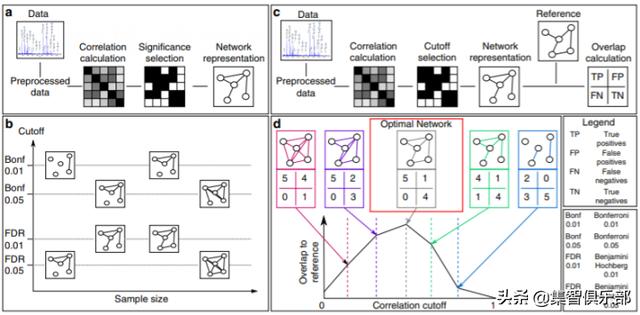 文章插图
文章插图
图3:两种重构方法工作流与效果对比
2.在具体网络中验证为了验证这一方法的可靠性 , 研究者在多种生物数据集上进行了实验 。 由于IgG糖组学已经被很好地描述 , 便于检验 , 所以研究者首先用它来测试网络推断方法的有效性 。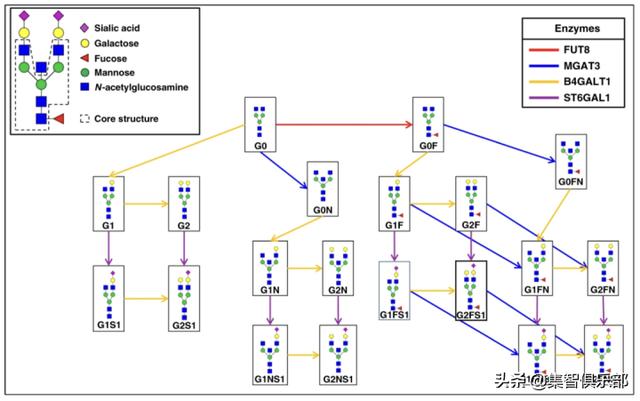 文章插图
文章插图
图4:免疫球蛋白G(IgG)聚糖结构的合成路径
以往研究已经较为清楚IgG糖基化的路径 , 如图4所示 , 节点代表聚糖结构 , 有向连边代表合成过程中的单个酶促反应 。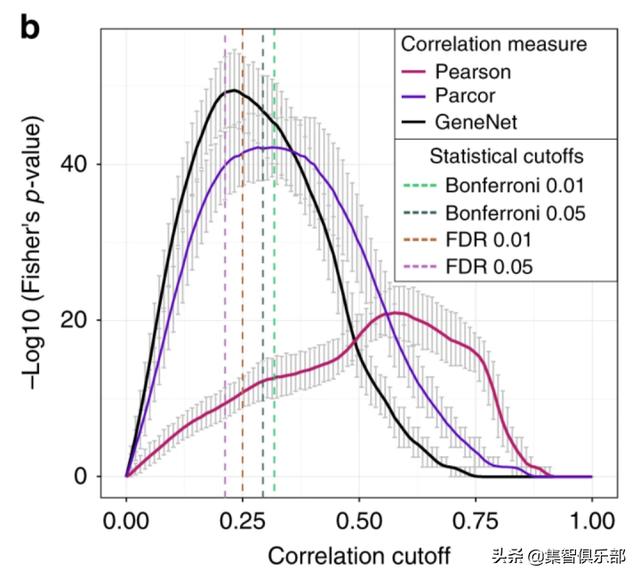 文章插图
文章插图
图5:IgG重构网络与参考网络重叠度的Fisher检验
利用该网络作为先验知识对具体网络进行重构 , 结果如图5 。 纵轴表示不同阈值下的Fisher检验的p值 , 该指标反映了被重构网络与参考网络的重叠程度 。 关联网络与生物学参考网络产生最大重叠时 , 称为最优截断(optimal cutoff) 。 实线的颜色代表了不同的相关系数确定方法 。 本文使用了三种相关性分析的方法 , Pearson相关性(粉色) , 精确的部分相关性(partial correlation , 称为parcor , 紫色)和基于GeneNet算法的部分相关性 。 图中的虚线则代表在原始数据矩阵上 , 利用GeneNet算法 , 以不同显著性衡量标准给出的截断(cutoff) , 即阈值 。 显然 , Pearson与parcor方法的效果并不好 , 而使用参考网络最优截断得到的最优的GeneNet网络非常接近GeneNet网络最优的统计截断点(虚线FDR=0.05) 。
- 余额|中兴通讯:现有资金余额仅能确保公司当前经营规模下现金流安全
- 科技成果|“基于第三代半导体光源的低投射比投影仪关键技术”通过科技成果评价
- Nat. Mater:它的出现是否能引领5G通讯的发展?
- 如何基于Python实现自动化控制鼠标和键盘操作
- 需要更换手机了:基于手机构建无人驾驶微型汽车
- 2021年 中兴通讯终端业务将全面回归中国2C市场
- 蓝鲸专访|水滴CTO邱慧:基于业务场景做技术创新,用户需求可分析并唤醒
- 中兴通讯高调官宣:统一操盘中兴、努比亚、红魔三大品牌
- GPU|干货|基于 CPU 的深度学习推理部署优化实践
- 怎么选择合适的无线通讯技术?
