使用CNN和Python实施的肺炎检测
介绍嘿!几个小时前我刚刚完成一个深度学习项目 , 现在我想分享一下我所做的事情 。 这一挑战的目标是确定一个人是否患有肺炎 。 如果是 , 则确定是否由细菌或病毒引起 。 好吧 , 我觉得这个项目应该叫做分类而不是检测 。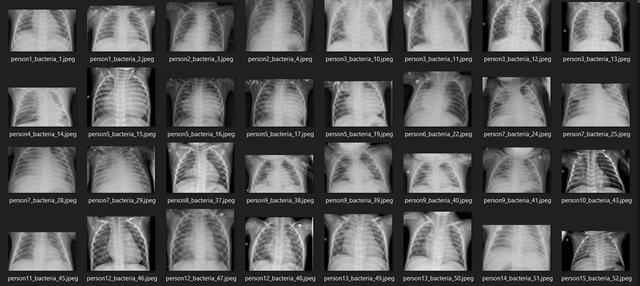 文章插图
文章插图
换句话说 , 此任务将是一个多分类问题 , 其中标签名称为:normal(正常) , virus(病毒)和bacteria(细菌) 。 为了解决这个问题 , 我将使用CNN(卷积神经网络) , 它具有出色的图像分类能力 ,。 不仅如此 , 在这里我还实现了图像增强技术 , 以提高模型性能 。 顺便说一句 , 我获得了80%的测试数据准确性 , 这对我来说是非常令人印象深刻的 。
整个数据集本身的大小约为1 GB , 因此下载可能需要一段时间 。 或者 , 我们也可以直接创建一个Kaggle Notebook并在那里编码整个项目 , 因此我们甚至不需要下载任何内容 。 接下来 , 如果浏览数据集文件夹 , 你将看到有3个子文件夹 , 即train , test和val 。
好吧 , 我认为这些文件夹名称是不言自明的 。 此外 , train文件夹中的数据分别包括正常 , 病毒和细菌类别的1341、1345和2530个样本 。 我想这就是我介绍的全部内容了 , 现在让我们进入代码的编写!
注意:我在本文结尾处放置了该项目中使用的全部代码 。
加载模块和训练图像使用计算机视觉项目时 , 要做的第一件事是加载所有必需的模块和图像数据本身 。 我使用tqdm模块显示进度条 , 稍后你将看到它有用的原因 。
我最后导入的是来自Keras模块的ImageDataGenerator 。 该模块将帮助我们在训练过程中实施图像增强技术 。
import osimport cv2import pickleimport numpy as npimport matplotlib.pyplot as pltimport seaborn as snsfrom tqdm import tqdmfrom sklearn.preprocessing import OneHotEncoderfrom sklearn.metrics import confusion_matrixfrom keras.models import Model, load_modelfrom keras.layers import Dense, Input, Conv2D, MaxPool2D, Flattenfrom keras.preprocessing.image import ImageDataGeneratornp.random.seed(22)接下来 , 我定义两个函数以从每个文件夹加载图像数据 。 乍一看 , 下面的两个功能可能看起来完全一样 , 但是在使用粗体显示的行上实际上存在一些差异 。 这样做是因为NORMAL和PNEUMONIA文件夹中的文件名结构略有不同 。 尽管有所不同 , 但两个功能执行的其他过程基本相同 。
首先 , 将所有图像调整为200 x 200像素 。
这一点很重要 , 因为所有文件夹中的图像都有不同的尺寸 , 而神经网络只能接受具有固定数组大小的数据 。
接下来 , 基本上所有图像都存储有3个颜色通道 , 这对X射线图像来说是多余的 。 因此 , 我的想法是将这些彩色图像都转换为灰度图像 。
# Do not forget to include the last slashdef load_normal(norm_path):norm_files = np.array(os.listdir(norm_path))norm_labels = np.array(['normal']*len(norm_files))norm_images = []for image in tqdm(norm_files):image = cv2.imread(norm_path + image)image = cv2.resize(image, dsize=(200,200))image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)norm_images.append(image)norm_images = np.array(norm_images)return norm_images, norm_labelsdef load_pneumonia(pneu_path):pneu_files = np.array(os.listdir(pneu_path))pneu_labels = np.array([pneu_file.split('_')[1] for pneu_file in pneu_files])pneu_images = []for image in tqdm(pneu_files):image = cv2.imread(pneu_path + image)image = cv2.resize(image, dsize=(200,200))image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)pneu_images.append(image)pneu_images = np.array(pneu_images)return pneu_images, pneu_labels声明了以上两个函数后 , 现在我们可以使用它来加载训练数据了 。 如果你运行下面的代码 , 你还将看到为什么我选择在该项目中实现tqdm模块 。
norm_images, norm_labels = load_normal('/kaggle/input/chest-xray-pneumonia/chest_xray/train/NORMAL/')pneu_images, pneu_labels = load_pneumonia('/kaggle/input/chest-xray-pneumonia/chest_xray/train/PNEUMONIA/') 文章插图
文章插图
到目前为止 , 我们已经获得了几个数组:norm_images , norm_labels , pneu_images和pneu_labels 。
带_images后缀的表示它包含预处理的图像 , 而带_labels后缀的数组表示它存储了所有基本信息(也称为标签) 。 换句话说 , norm_images和pneu_images都将成为我们的X数据 , 其余的将成为y数据 。
为了使项目看起来更简单 , 我将这些数组的值连接起来并存储在X_train和y_train数组中 。
X_train = np.append(norm_images, pneu_images, axis=0)y_train = np.append(norm_labels, pneu_labels)
- OPPO|OPPO未来科技大会正式官宣,官方明示将有旗舰新品和创新技术亮相
- 小米科技|雷军:小米12即将发布!首款搭载骁龙8手机,性能和功耗咋样?
- 支付宝|突破2项关键技术,中科院又立功了,事关量子计算和3D打印
- 闪存|变频器要怎样使用才能确保省电?
- |联想小新air和pro的区别大吗?哪个性能更强?详细解读
- 荣耀|建议收藏!2021年底盘点:这三款旗舰可以让你安逸地使用两三年
- 英伟达|Linux下使用KVM虚拟机安装华为OpenEuler系统
- 主板|华为智慧屏视频通话功能怎么使用,操作难不难?
- |LoRa做到了!实现低功耗和远距离通信!
- 监管机构|谷歌和Meta被俄罗斯监管机构告上法庭,或面临巨额罚款
